各種in silicoスクリーニング、シミュレーションによる検証、化合物データベース整理、最適なソフトウェア・システム導入など、優れた費用対効果でトータルに創薬研究をサポートします。
こんな課題を解決します
- タンパク質の立体構造から、活性部位に結合する化合物をデザインしたい
- 手持ちのリガンドを元に、より活性の高い化合物を見つけたい
- スクリーニングで見つけたリガンドの標的タンパク質を予測したい
4つのin silico手法で創薬を支援
| 計算に必要な条件・データなど | 計算手法と結果の特徴 | 新規骨格 | 計算時間 | 標的予測 | |
|---|---|---|---|---|---|
| SBVS ドッキングシミュレーション |
・標的タンパク質の立体構造 ・既知化合物との共結晶構造が望ましい ・類縁構造が既知の場合、ホモロジーモデリングで準備可能 |
・標的タンパク質のポケットと化合物の結合様式をシミュレートする ・新規構造の化合物の探索に有効 |
◯ | △ | ✕ |
| PBVSファーマコフォアベース法 | ・リガンドベース構築の場合: 既知活性化合物の情報(数個程度) ・構造ベース構築の場合: 標的タンパク質とリガンドの共結晶構造 (PDB) |
・活性化合物からファーマコフォアモデルを作成して候補化合物を絞り込む ・複合体構造情報からファーマコフォアモデルを作成することも可能 |
◯ | ◎ | ◯ |
| LBVS 類似化合物探索法(QSAR等を含む) |
・既知活性化合物の情報(数個〜) ・標的タンパク質が明確でない場合も有効 |
・既知活性化合物に対する類似性を指標として化合物を探索する ・既知構造の周辺化合物の探索に有効 ・新規構造の化合物の探索には不向き |
✕ | ◎ | ◯ |
| CGBVS 相互作用マシンラーニング法 |
・標的タンパク質の一次配列情報 ・既知活性、不活性化合物の情報 例)IC50<30μMのリガンドデータ100個 |
・予測モデル作成に学習用活性データセットが必要 ・膨大な既知データの機械学習によるパターン認識に基づいて相互作用を予測する ・標的タンパク質の周辺(類縁)タンパク質の既知活性情報も有効に活用できる |
◯ | ◎ | ◯ |
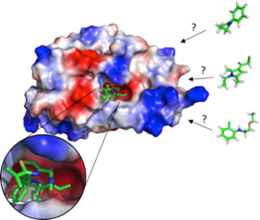
ドッキングシミュレーション法(SBVS)
標的タンパク質の立体構造モデルを用いて、活性ポケットに結合する化合物を探索します。
ファーマコフォアベース法(PBVS)
リガンドの官能基特性(電荷、水素結合のドナー/アクセプタ、疎水性領域、芳香環など)を元にして三次元モデル(ファーマコフォアモデル)を構築し、新規化合物探索に用います。 モデルの構築には、既知の活性化合物の高精度アライメント(リガンドベース)またはタンパク質立体構造との複合体の活性コンフォメーションの利用(構造ベース)、いずれかの方法を用います。
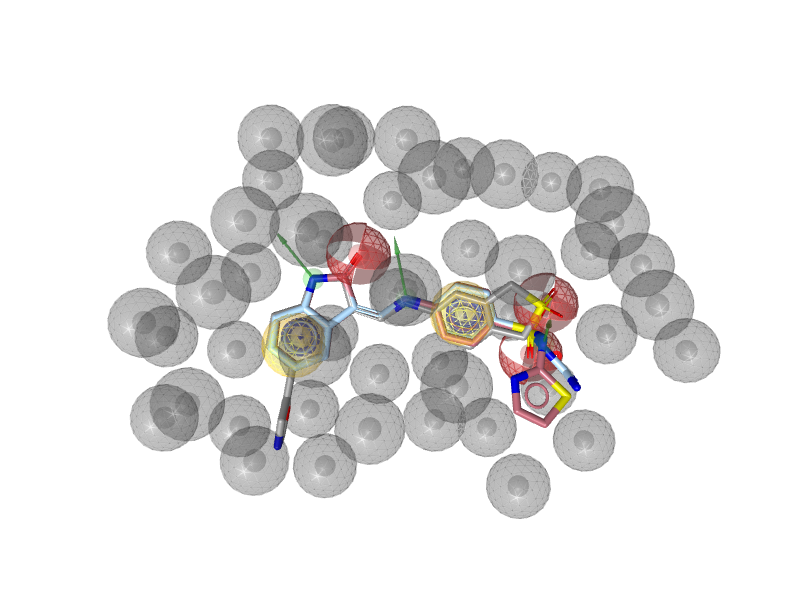
リガンドベースのファーマコフォアモデル
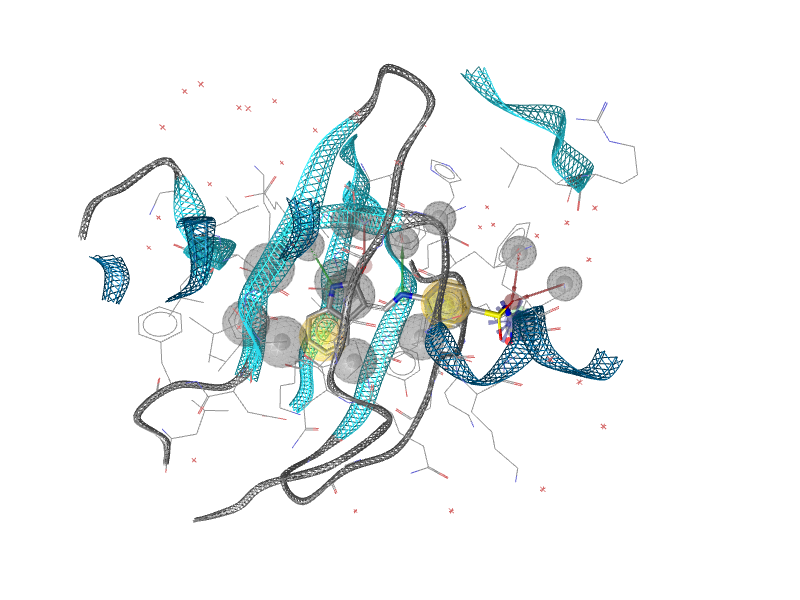
構造ベースのファーマコフォアモデル
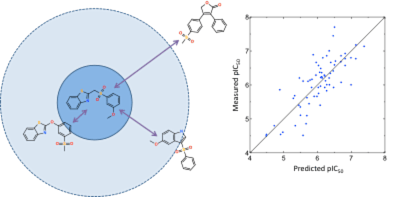
類似化合物探索法(LBVS)
分子構造や物性に基づいて、既知の活性化合物に類似する化合物を探索します。
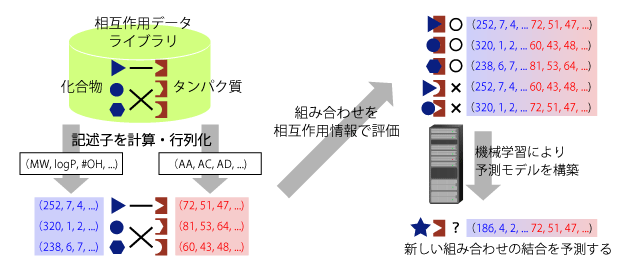
相互作用マシンラーニング法(CGBVS)
低分子化合物を分子量や極性、分配係数、各種官能基の数など3000〜4000次元の行列で、タンパク質のアミノ酸配列も行列で表して、既知の相互作用がある組み合わせに共通するパターンを機械学習します。この学習結果を用いて、新規の化合物についての相互作用予測や、標的タンパク質の予測を行います。
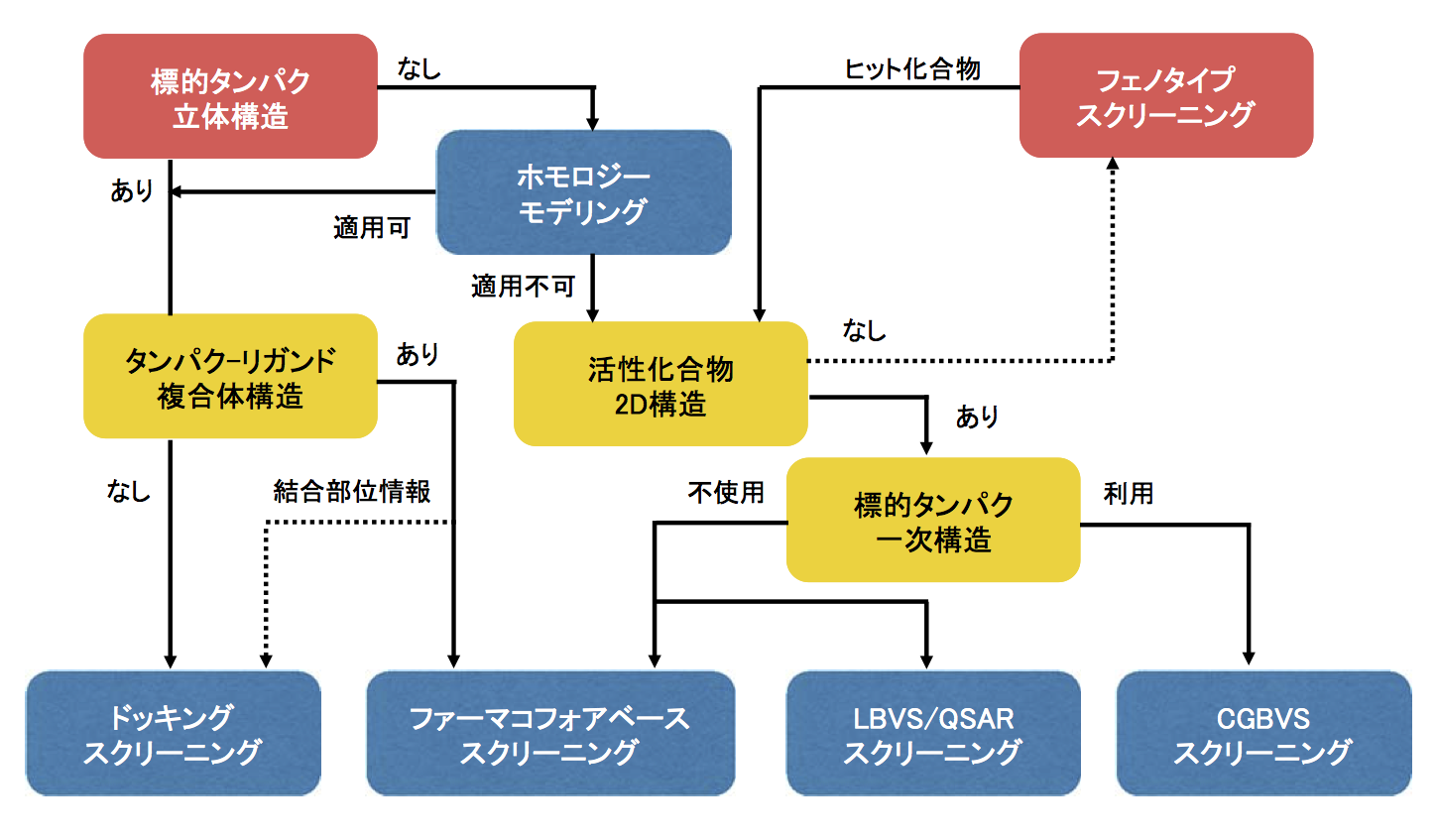
最適な手法は?
基本的には、下記のチャートのように判断ができます。ご不明な点は、ぜひご相談ください。
価格
基本料金+個別計算料にて承っております。
個別計算料につきましては、都度お見積りいたします。
基本料金:400,000円(税別)
個別計算料の例(すべて税別)
| 計算メニュー | 個別計算料 |
|---|---|
| CGBVS 化合物探索計算 500万化合物 | 720,000円 |
| CGBVS 標的タンパク質予測計算 10化合物 | 360,000円 |
| CGBVS 予測モデル構築 | 720,000円 |
| de novoデザイン(CGVBS×PSO/GA) | 1,800,000円 |
| 高速ファーマコフォア 500万化合物 | 720,000円 |
| ファーマコフォア 1万化合物 | 360,000円 |
| 高速ドッキングスクリーニング 100万化合物 | 720,000円 |
| ドッキングスクリーニング 1000万化合物 | 360,000円 |
| 精密ドッキング解析 10化合物 | 360,000円 |
| ホモロジーモデリング 鋳型配列同一性 40%以上 | 360,000円 |
| リガンドベーススクリーニング 500万化合物 | 360,000円 |